【摘要】 分子模拟教程,VMD统计并绘制氢键
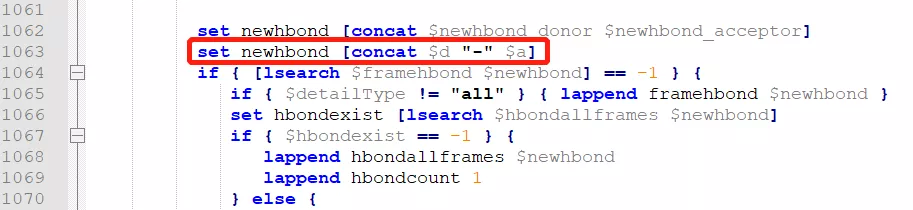
1)修改hbonds插件源代码输出原子编号
E:\Program Files(x86)\VMD\plugins\noarch\tcl\hbonds1.2\hbonds.tcld的hbonds插件源代码,在1062行后添加一行set newhbond [concat $d "-" $a],将输出改为每个氢键涉及的氧原子,然后保存。
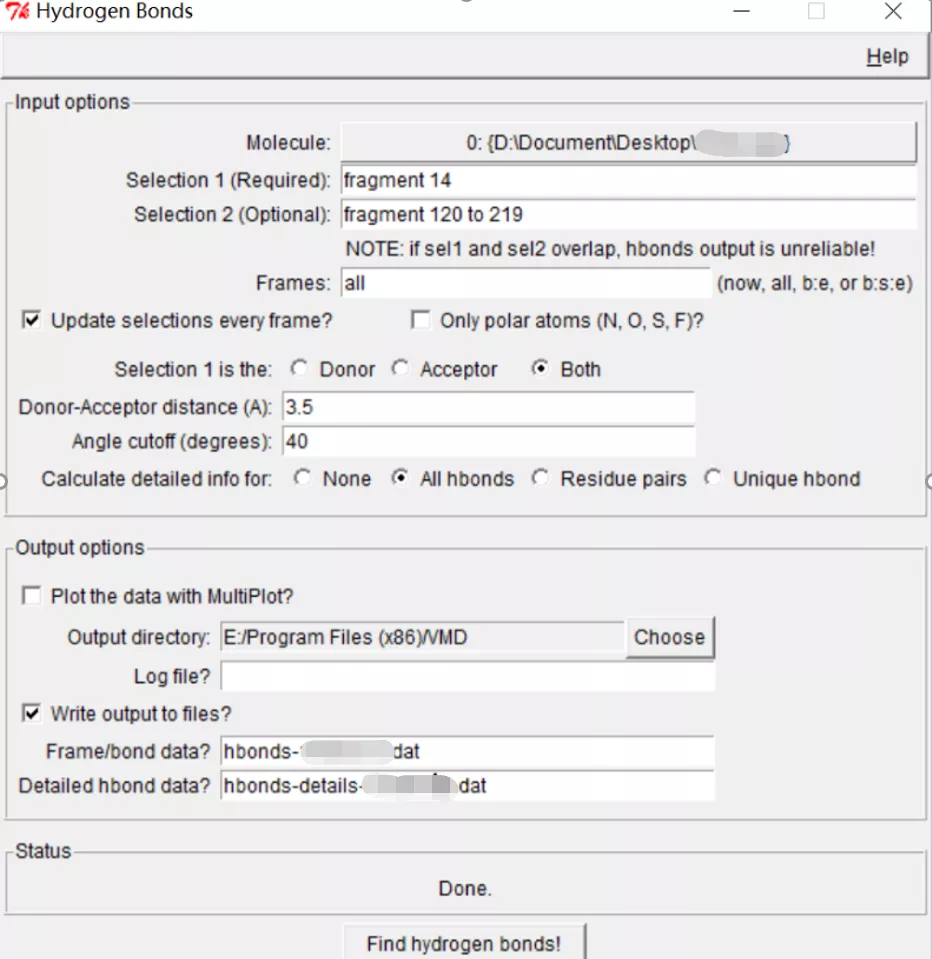
2)获取轨迹的氢键原子编号
Extension-analysis-Hydrogen Bonds,设置参数,selection是根据.gro文件中选择相应的原子(从0开始,需要减1)。
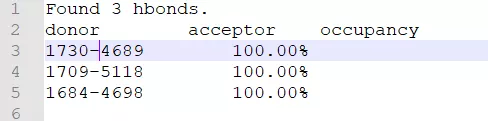
hbonds.dat: 每帧的氢键数目,第一列为帧号,第二列为氢键数目。
hbonds-details.dat: 所有氢键的供体,受体,占有率,内容类似如下:
3)将氢键原子编号整理为选择语句
将前一步得到的hbonds-details.dat中的氢键原子编号整理为选择语句,类似index 1730 or index 4689 or index 1709 or index 5118 or index 1684 or index 4698,即所有可能参与分子间氢键形成的氧原子的编号。
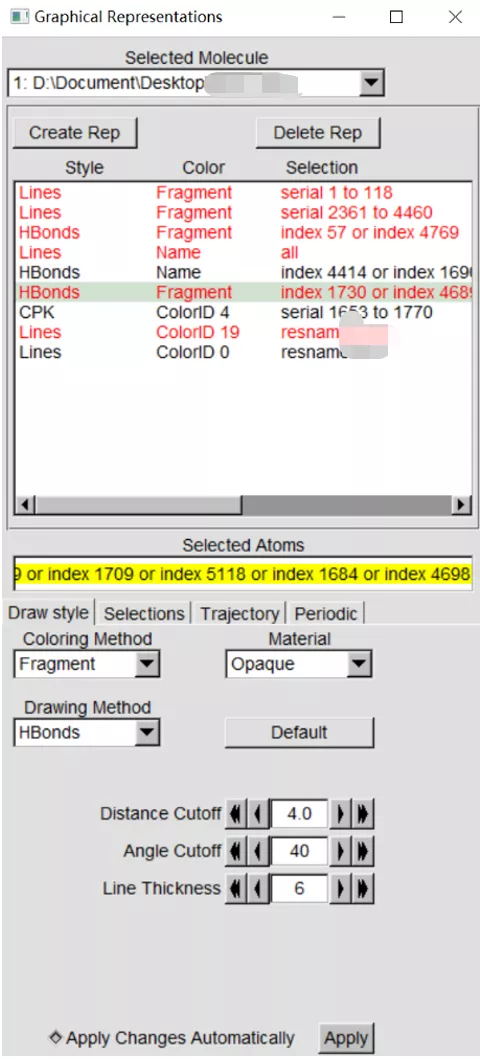
4)使用氢键模式显示选择的原子
以前一步的选择语句创建新的rep,并以HBonds模式显示。
5)统计两个原子之间的距离
在VMD窗口活动的情况下在小键盘上点击测定键长的快捷键“2”,依次点击需要确定距离的两个原子;
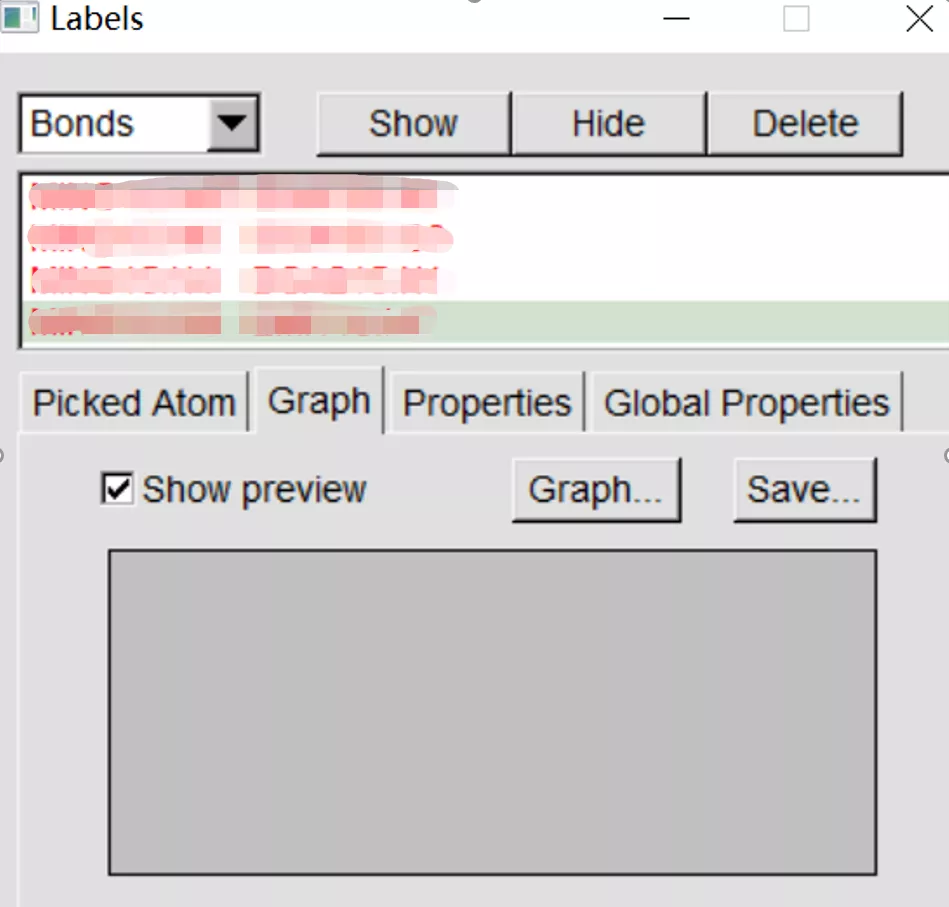
在VMD主菜单中的Graphics-labels;
将默认显示为“Atoms”的下拉菜单改成“Bonds”,此时,被测定的两个原子的信息会显示在显示框内,如“SER 387:CB SER387:OG”;
点击对话框中的“Graph”,可以看见“Graph”和“Save”两种选择:点击前者会显示距离图,点击后者会出现保存距离信息的提示。
参考链接:https://jerkwin.github.io/











 您已经拒绝加入团体
您已经拒绝加入团体

 2021-12-06
2021-12-06
 5664
5664
 1
1











