【摘要】 最近小编回顾了一篇古早的JACS,并决定以文中报道的四金属中心开壳层体系为例,聊聊如何绘制自旋密度等值面图并计算自旋布居。
最近小编回顾了一篇古早的JACS,并决定以文中报道的四金属中心开壳层体系为例,聊聊如何绘制自旋密度等值面图并计算自旋布居。
早在2000年,法国Architecture et Fonction des Macromolécules Biologiques, Christian Cambillau教授课题组报道并解析了分离自Pseudomonas nautica (Pn)的氧化亚氮还原酶(Nitrous oxide reductase, N2OR)某亚基中的μ4-Sulfide Bridged Tetranuclear CuZ结构,如下图所示[1]。
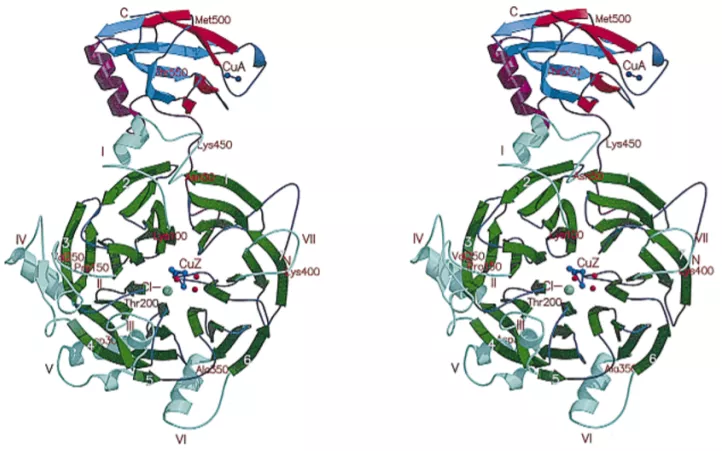
Fig.1 Stereo view of the crystal structure of the N2OR monomer
自然界中,N2OR在无氧代谢的最终步催化了温室气体N2O的双电子还原,生成N2与H2O,并参与了ATP合成[2]。而2002年,斯坦福大学化学系Monroe E. Spaght Professor of Chemistry, Edward I. Solomon课题组博士研究生Peng Chen(现为康奈尔大学化学与化学生物学系Peter J. W. Debye Professor of Chemistry)进一步通过磁圆二色柱、X射线吸收光谱、电子顺磁共振以及模拟计算密度泛函理论等方法进一步研究了该体系内氧化态、自旋状态、基态波函数等性质[3]。
近20年后的今天,小编决定依靠现有的方法与基组效仿这篇JACS,研究这一Cuz中心体系的自旋密度与自旋布居。值得一提的是,自然界中这样水环境中的四铜中心往往与多个组氨酸进行配位,为了简化问题,本文以氨分子为模型代替组氨酸研究上述问题。
01结构优化与频率计算
我们首先在GaussView中画出如下的类四面体结构(Cuz_NH3,下同)进行结构优化与频率计算,关键字为:
# opt freq em=gd3bj pbe1pbe/def2sv
.webp)
02导出Gaussian-type格点文件
我们将得到的.chk文件(checkpoint file)转换为Gaussian Formatted Checkpoint (.fchk)文件,并用Multiwfn (_3.7)读取(谈谈自旋密度、自旋布居以及在Multiwfn中的绘制和计算 sobereva.com/353)。以绘制三维坐标下等值面图为例,在Multiwfn主功能菜单中,我们依次键入:
5 //Output and plot specific property within a spatial region (calc. grid data);
5 //Electron spin density;
3 //High quality grid, covering whole system, about 1,728,000 points in total;
2 //Export data to a Gaussian-type cube file in current folder。
这样一来我们就得到了在同一路径下的.cub文件(spindensity.cub)。
03绘制自旋密度等值面图
随后我们打开VMD程序(http://www.ks.uiuc.edu/Research/vmd/)绘制Cuz_NH3的自旋密度等值面图。此前小编已经将Multiwfn程序包中examples\scripts目录下的VMD脚本showcub.vmd以及上一步我们导出的cube文件spindensity.cub拷贝至VMD目录下(VMD里将cube文件瞬间绘制成效果极佳的等值面图的方法 sobereva.com/483)。
在VMD主界面输入source showcub.vmd,Enter后输入cub + 文件名“spindensity”(注意此时不要输入后缀“.cub”)再次Enter(以windows系统为例,如果.cub文件在别的文件夹下,键入路径时切记将“\”改写为“\\”)。此时我们将立即看到下图VMD生成的Default可视化结构(此时分辨率较低的问题我们将在下文解决)。
.webp)
我们进一步优化绘制出效果更好的等值面图像:
1.在VMD Main界面中Display-Depth Cueing不勾选,Display-Axes勾选off,Display-Rendermode中选择GLSL,Graphics-Colors-Display-Background中选择#8 white;
2.Graphics-Colors按Element着色,元素颜色根据个人喜好选择,本例中选择Cu #21 cyan2, S #04 yellow, N #00 blue, O #01 red, H #08 white。
3.Graphics-Representations下我们一共create 7个Rep:
a)第1个Rep中,Coloring Method选择Element,Drawing Method选择CPK;
b)第2、3个Rep中,Drawing Method均选择Isosurface(Draw-Solid Surface, Show-Isosurface),Material可根据个人喜好,本例选择Transparent。而在两Rep中Isovalue分别输入0.002与-0.002,Coloring Method-ColorID分别选择#01 red与#04 yellow。本例中因为没有自旋密度为负的部分,故Isovalue为负值的Rep无法可视化体现;
c)第4-7个Rep中,Coloring Method选择Element,Material本例选择Opaque,Drawing Method选择Dynamic Bonds。Selected Atoms中分别手动键入element Cu Cu(或只写“element Cu”,显示Cu-Cu键,下同)、element Cu S、element Cu N、element Cu O(Bond Radius均为0.1,Bond Resolution均为12)。其中Cu-Cu键Distance Cutoff 1.6,Cu-S键Distance Cutoff 2.3,Cu-N键Distance Cutoff 2.1,Cu-O键Distance Cutoff 3.1。
所有设定结束后,我们得到下图:
.webp)
我们注意到:
1.SCu4结构并非严格的(类)四面体结构,可能由于水分子的配位导致Cu-Cu间距的不同,本例中并非所有Cu原子两两间均成键;
2.未成对电子主要围绕着S及Cu原子分布,似乎集中在S原子的未成键区域,并在Cu原子周围以d轨道形状分布,在N原子周围亦有分布。
为了提高分辨率,我们在VMD main-File-Render一栏中,Render the current scene using:选择Tachyon,并在Render Command的末尾(bmp)后手段键入“-res 1500 1500”进行render。
04Multiwfn进行自旋布居分析
为了定量说明“注意#02”,我们继续使用Multiwfn进行自旋布居分析。将.fchk文件拖入Multiwfn后,在主功能菜单中依次键入:
7 //Population analysis and atomic charges;
5 //Mulliken atom & basis function population analysis;
1 //Ouput Mulliken population and atomic charges.
此时Multiwfn输出的Population of atoms一栏中Spin Pop.一列即为各原子的自旋布居,如下图所示。
.webp)
可见,体系总共1个单电子,其中~0.86个集中在S原子和Cu原子上。而上拉至Population of each type of angular moment atomic orbitals一栏,我们可以发现S原子的p轨道贡献了0.31个单电子,而四个Cu原子的d轨道各有0.1-0.2的贡献,在N上只有极少量单电子。
.webp)
References:
[1] Brown, K.; Tegoni, M.; Prudencio, M.; Pereira, A. S.; Besson, S.; Moura, J. J.; Moura, I.; Cambillau, C. Nat. Struct. Biol. 2000, 7, 191;
[2] Zumft, W. G. Microbiol. Mol. Biol. Rev. 1997, 61, 533;
[3] Chen, P.; George S. D.; Cabrito, I.; Antholine, W. E., Moura, J. J. G.; Moura, I.; Hedman, B.; Hodgson, K. O.; Solomon, E. I. J. Am. Chem. Soc. 2002, 124, 744.











 您已经拒绝加入团体
您已经拒绝加入团体

 2021-07-29
2021-07-29
 4887
4887
 0
0










